Un algoritmo genético es una búsqueda heurística que está inspirada en la teoría natural de la evolución de Darwin. Este algoritmo refleja el proceso de selección natural, donde los individuos más aptos son seleccionados para reproducirse en orden, para producir descendencia de la siguiente generación.
Esta noción de selección natural puede ser aplicado para resolver problemas. Consideramos que un grupo de soluciones para un problema y seleccionamos al mejor grupo de soluciones de todos ellos.
Cinco faces son consideradas en un algoritmo genético.
U individuo es caracterizado por un grupo de parámetros (variables) conocidos como Genes. Los genes son agrupados en cadena para forman un Chromosome (solución)
En un algoritmo genético, el grupo de genes y un individuo son representados usando una cadena. Usualmente son usados valores binarios. Decimos que codificamos los genes en un cromosoma.
Esta función determina como evaluar a un individuo (La habilidad de un individuo para competir con otros individuos). Entrega un fitness score para cada individuo. La probabilidad que un individuo será seleccionado para reproducirse está basado en el fitness score.
La idea de la fase de selección, es para seleccionar al individuo más apto y dejar que pase sus genes a la siguiente generación.
Dos pares de individuos (padres) son seleccionados basado en su fitness score. Los individuos con altos fitness tienen más probabilidad de ser seleccionados para reproducirse.
Es la fase más importante del algoritmo genético. Para cada par de padres que seran emparejados, un punto de crossover es seleccionado de forma aleatoria dentro de los genes.
La nueva descendenca es aderida a la población.
La mutación ocurre para mantener la diversidad dentro de la población y prevenir una convergencia prematura.
El algoritmo termina si la población converge (ya no se producen más descendencia con diferencias significativas entre ellas). Entonces se dice que el algoritmo genético ha provisto de un grupo de soluciones a nuestro problema.
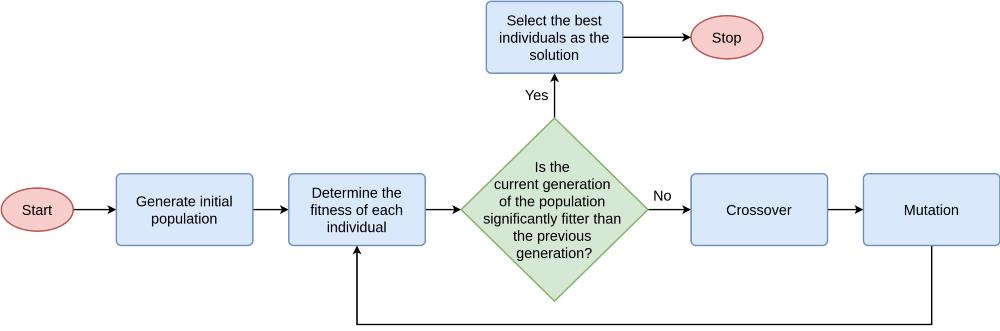
La siguiente figura muestra el diagrama de flujo de como trabaja un algoritmo genético.
En nuestro ejemplo una institución necesita crear un horario, con la mínima cantidad de cruces posibles, además solo cuenta con un profesor para cada Idioma.
Se requiere que se programen 10 idiomas con dos secciones cada una A y B con dos sesiones de dos horas de clases a la semana de cada sección e idioma, teniendo en cuenta que solo se trabaja de 19 a 23 de la noche, de lunes a sábados.
Consta de individuos con la siguiente distribución de genes.
GEN1 | GEN2 | GEN3 | GEN4 | GEN5 | GEN6 | ... | GEN40
IDIOMA XYZ ==> Se repite hasta completar con los 10 idiomas
Debido a las condiciones del problema, sabemos que solo existen 12 bloques de horas en la semana para programar nuestros horarios. Por ello podemos codificar el 12 en binario. Dando como resultado que cada GEN esté compuesto por 4 bits.
Tomaremos como criterio de más apto a aquel en dónde los cruces sean menores y sobre todo a aquello en dónde no existan cruces en mismo idioma.
Se seleccionan al 70% superior de toda la población y se eliminan al resto, luego se completa la población repitiendo aleatoriamente a estos 70% para completar la población de padres.
Se toman como puntos de crossover a los incios y finales de cada gen dentro de los cromosomas padres para realizar intercambios de genes.
Dentro de cada cromosoma se elige de forma aleatoria un bit que será cambiado de estado, este proceso de cambio aleatorio lo podemos repetir tantas veces como configuremos el factor de mutación.
def crear_poblacion(cant_poblacion, cant_horas,cant_dias,cant_idiomas,cant_secc,cant_sesiones):
cant_genes = cant_idiomas*cant_secc*cant_sesiones
cant_bin_per_gen = 0
for num in [cant_horas*cant_dias]:
cant_bin_per_gen += len(str(bin(num))[2:])
temp_pobl = np.random.random_integers(low= 0, high=1,size=(cant_poblacion,cant_bin_per_gen*cant_genes) )
return temp_pobl,cant_bin_per_gen
def decodificador(poblacion,cant_horas,cant_dias,cant_idiomas,cant_secc,cant_sesiones,cant_bin_per_gen):
def sess_bin_dec(num,cant_sessiones_unicas):
"""
num es un vector = [1,0,1,0,...,1] que representa un número binario
cant_sessiones_unicas = valor que consiste en multiplicar HORARIOS DISPONIBLES POR DÍA * DÍAS QUE SE PUEDEN DICTAR
"""
mult = np.stack([2**(i) for i in range(len(num))][::-1])
dec = np.dot(num,mult)
while dec>=cant_sessiones_unicas:
dec = dec%cant_sessiones_unicas if dec>=cant_sessiones_unicas else dec
return dec
cant_genes = cant_idiomas*cant_secc*cant_sesiones
cant_sessiones_unicas = cant_horas*cant_dias
num_individuos = poblacion.shape[0]
poblacion_deco = np.zeros((num_individuos,cant_genes))
for ind, individuo in enumerate(poblacion):
poblacion_deco[ind] = [sess_bin_dec(individuo[ini_bin:(ini_bin+cant_bin_per_gen)],cant_sessiones_unicas) for ini_bin in range(cant_genes)]
return poblacion_deco[0]
def eval_poblac_fitness(poblacion,cant_horas,cant_dias,cant_idiomas,cant_secc,cant_sesiones,cant_bin_per_gen):
"""
Se han reconocido 3 tipos de cruces de horarios
1) Cruces entre secciones - mismo curso = x1000
2) Cruces entre secciones - diferenctes curso = x1
3) Cruces entre sesiones del mismo curso = x100
Función de evaluación:
f(x1 + x2 + x3) = -(x1*1000+x2+x3*100)
## EVALUACIÓN
1 - (100)comprobar traslape entre seSSiones del mismo curso =>> verificar que los casilleros de las seSSiones sean diferentes | Dentro de la misma sección A o B de cada curso, comprobar que las dos sesiones dentro de la sección A o B sean diferentes.
EJEMP: La sesión1 de la semana se traslapan con la sesión2 de la semana
2 - (100)comprobar traslape entre seCCiones del mismo curso =>> verificar que los casilleros de las seSSiones sean diferentes | Agrupando cada seCCión A y B para un mismo curso, comprobar similitud de casillero de sesión entre A y B del mismo curso
EJEMP: La sección A y B del español se traslapan
3 - (1) comprobar traslape entre seCCiones de DIFERENTE curso =>> verifivar que los casilleros de las seSSiones sean diferentes | Agrupando cada curso con su sección A y B, para compararla con la de otro
EJEMP: La sección AóB del español se traslapa con la sección AóB del Ingles en alguna sesión de la semana
"""
# Función auxiliar para trasnformar un gen en un número de casillero de sesión en el calendario
def sess_bin_dec(num,cant_sessiones_unicas):
"""
num es un vector = [1,0,1,0,...,1] que representa un número binario
cant_sessiones_unicas = valor que consiste en multiplicar HORARIOS DISPONIBLES POR DÍA * DÍAS QUE SE PUEDEN DICTAR
"""
mult = np.stack([2**(i) for i in range(len(num))][::-1])
dec = np.dot(num,mult)
while dec>=cant_sessiones_unicas:
dec = dec%cant_sessiones_unicas if dec>=cant_sessiones_unicas else dec
return dec
# Evalua el criterio 1
# def determinar_igualdad_session(hr1,hr2):
# return hr1==hr2
# vf_horario = np.vectorize(determinar_igualdad_session)
# Evalua el criterio 2 incluye a la evaluación 1
def determinar_igualdad_session_secc(hr1,hr2,hr3,hr4):
temp = [hr1,hr2,hr3,hr4]
igualdad = [hr_p == hr_q for ind, hr_p in enumerate(temp[:-1]) for hr_q in temp[ind+1:]]
return sum(igualdad)
vf_horario_secc = np.vectorize(determinar_igualdad_session_secc)
# Evalua el criterio 3
# DECODIFICANDO CROMOSOMA
cant_genes = cant_idiomas*cant_secc*cant_sesiones
cant_sessiones_unicas = cant_horas*cant_dias
num_individuos = poblacion.shape[0]
num_num_bin = poblacion.shape[1]
poblacion_deco = np.zeros((num_individuos,cant_genes))
for ind, individuo in enumerate(poblacion):
poblacion_deco[ind] = [sess_bin_dec(individuo[ini_bin:(ini_bin+cant_bin_per_gen)],cant_sessiones_unicas) for ini_bin in range(cant_genes)]
#print(poblacion_deco)
## Evaluando el primer tipo de cruce | traslape entre sesiones de una misma sección
# agrupado_sess = np.reshape(poblacion_deco, (cant_idiomas,-1,cant_sesiones))
# eval_1 = np.sum(vf_horario(agrupado_sess[:,:,0],agrupado_sess[:,:,1]), axis=1)
## Evaluando el segundo tipo de cruce | traslape entre secciones de un mismo curso
agrupado_idioma_sess = np.reshape(poblacion_deco, (num_individuos,-1,cant_sesiones*cant_secc))
eval_2 = np.sum(vf_horario_secc(agrupado_idioma_sess[:,:,0],agrupado_idioma_sess[:,:,1],agrupado_idioma_sess[:,:,2],agrupado_idioma_sess[:,:,3]), axis=1)
## Evaluando el tercer tipo de cruce | traslape entre secciones de diferentes cursos
eval_3 = []
for ind, individuo_x in enumerate(agrupado_idioma_sess):
eval_ = 0
for i, idioma_i in enumerate(individuo_x[:-1]):
for idioma_j in individuo_x[i+1:]:
for sess_i in idioma_i:
eval_+=sum(sess_i == idioma_j)
#print(eval_)
eval_3.append(eval_)
eval_3 = np.stack(eval_3)
## Computando la evaluación general de cada individuo
eval_total = -(eval_2*100 + eval_3)
#print(vf_horario(agrupado_sess[:,:,0],agrupado_sess[:,:,1]))
#return np.sum(vf_horario(agrupado_sess[:,:,0],agrupado_sess[:,:,1]), axis=1)
return eval_total , np.max(eval_total)
def seleccion(poblac_init,fitness_eval_result,threshold_padres):
#print(fitness_eval_result)
padres = poblac_init.copy()
fitness_eval = fitness_eval_result.copy()
num_individuos = poblac_init.shape[0]
num_num_bin = poblac_init.shape[1]
#Se reemplaza el peor cromosoma por el mejor
poblac_init[np.argmin(fitness_eval_result)]=poblac_init[np.argmax(fitness_eval_result)]
fitness_eval[np.argmin(fitness_eval_result)]=fitness_eval[np.argmax(fitness_eval_result)]
#Se ordenan los resultados (mayor=>menor)
fitness_sort = np.sort(fitness_eval)[::-1]
#Se escoge a los X primeros
n_primeros = round(num_individuos*(100-threshold_padres)/100)
best_n = fitness_sort[:n_primeros]
#print(best_n)
for ind in range(num_individuos):
if not fitness_eval[ind] in best_n:
ind_selected = np.where(fitness_eval == random.choice(best_n))[0][0]
#print(ind_selected)
padres[ind] = poblac_init[ind_selected ]
fitness_eval[ind] = fitness_eval[ind_selected]
return padres
def crossover(padres,cant_bin_per_gen,cant_genes):
num_individuos = padres.shape[0]
num_num_bin = padres.shape[1]
e=0
puntos_crossover=np.array([cant_bin_per_gen*2*i for i in range(cant_genes//2)])[1:]
#print(puntos_crossover)
auxiliar=np.zeros((num_individuos,num_num_bin))
for i in range(0,num_individuos,2):
#print(i)
hijo1=np.append(padres[i ][:puntos_crossover[e]] , padres[i+1][puntos_crossover[e]:])
hijo2=np.append(padres[i+1][:puntos_crossover[e]] , padres[i ][puntos_crossover[e]:])
auxiliar[i]=hijo1
auxiliar[i+1]=hijo2
e=e+1
return auxiliar
def mutacion(crossover,factor_mutacion):
mutado = crossover.copy()
num_individuos = crossover.shape[0]
num_num_bin = crossover.shape[1]
for _ in range(factor_mutacion):
for i in range(num_individuos):
pos_aleatoria=random.randrange(0,num_num_bin)
mutado[i][pos_aleatoria] = 1- mutado[i][pos_aleatoria]
return mutado
def stop_criterio(poblacion, fitness_eval_result,threshold_val,memoria_best_eval, threshold_std_eval):
curr_max_eval = np.max(fitness_eval_result)
memoria = memoria_best_eval.copy()
memoria.sort()
if len(memoria)>1000:
memoria = np.stack(memoria[-10:])
memoria_std = np.std(memoria)
if memoria_std <= threshold_std_eval:
print("Criterio de la desviación Estandar")
return True
if threshold_val <= curr_max_eval:
print("Criterio de valor óptimo menor a {}".format(threshold_val))
return True
def print_eval_result():
return 0
def print_producto(poblacion, fitness_eval_result,cant_horas,cant_dias,cant_idiomas,cant_secc,cant_sesiones,cant_bin_per_gen,HORARIOS):
tabla = pd.DataFrame(np.zeros((cant_horas,cant_dias)))
tabla.columns = ['LUNES', 'MARTES', 'MIERCOLES', 'JUEVES', 'VIERNES', 'SABADO']
IDIOMAS = ['ING', 'ALM', 'FRAN', 'ITA', 'PORT', 'CH','RS', 'JAP', 'ARAB', 'ESP' ]
tabla = tabla.replace(0,'')
#print(tabla)
curr_max_eval = np.max(fitness_eval_result)
best_individuo = poblacion[np.where(fitness_eval_result == curr_max_eval)]
best_individuo = decodificador(best_individuo,cant_horas,cant_dias,cant_idiomas,cant_secc,cant_sesiones,cant_bin_per_gen)
best_individuo_ind = np.reshape(best_individuo, (cant_idiomas,-1))
#best_individuo = np.stack([HORARIOS[int(i)] for i in best_individuo])
#best_individuo = np.reshape(best_individuo, (cant_idiomas,-1))
prefix = ''
for idioma, ind in enumerate(best_individuo_ind):
#print(ind)
#print('===================')
prefix = IDIOMAS[idioma]
for secc, pos in enumerate(ind):
if secc == 0 or secc == 1:
prefix_secc = prefix +'-A'
else:
prefix_secc = prefix + '-B'
if pos< cant_dias:
#print(pos)
tabla.iloc[0,int(pos)] = tabla.iloc[0,int(pos)] + prefix_secc + HORARIOS[0]+'\n'
else:
#print(int(pos-cant_dias))
tabla.iloc[1,int(pos-cant_dias)] = tabla.iloc[1,int(pos-cant_dias)] + prefix_secc + HORARIOS[1]+'\n'
tabla.to_csv(path_or_buf= 'best.csv', index=False )
print("Mejor Individuo: ")
print(tabla)
print("Mejor Puntaje")
print(curr_max_eval)
Un algoritmo genético al ser un proceso aleatorio en su naturaleza depende mucho de los valores iniciales y los espacios de tiempo aleatorio, es por ello que no siempre se obtienen los mismos resultados con la misma cantidad de iteraciones, sin embargo, es un algoritmo muy útil cuando se necesita atacar problemas complejos sin la necesidad de utilizar un procedimiento complejo.
Tengo un canal en youtube dónde tengo publicado un video completo sobre este tópico, me pueden buscar allí y aprender más cómo David Castillo.